概要
Pythonで機械学習をするときに使うscikit-learn(恐らく、サイキットラーンと呼びます)には前処理用のライブラリであるpreprocessingが組み込まれています。前処理とは、データの欠損保管や正規化、カテゴリデータの数値化等のことです。このpreprocessingの全メソッドの効果を検証したのが今回の記事です。irisのデータを題材にして説明していきます。
#preprocessingはspという名前で使うこととする
import sklearn.preprocessing as sp
#お決まりのライブラリをインポート
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
if __name__ == "__main__":
#irisデータをdfに格納
df = pd.read_csv("iris.csv", index_col=0)
メソッド一覧
コンソール上でdir(sp)と入力すると、メンバの一覧が取得できます。その中でメソッドを抽出すると以下23メソッドがあることが分かります。以降の項目で各メソッドについて説明していきます。
| add_dummy_feature | binarize | Binarizer | data |
| FunctionTransformer | imputation | Imputer | KernelCenterer |
| label | LabelBinarizer | LabelEncoder | maxabs_scale |
| MaxAbsScaler | minmax_scale | MinMaxScaler | MultiLabelBinarizer |
| normalize | Normalizer | OneHotEncoder | PolynomialFeatures |
| robust_scale | scale | StandardScaler |
add_dummy_feature
項目を1つ追加したいときに使う。
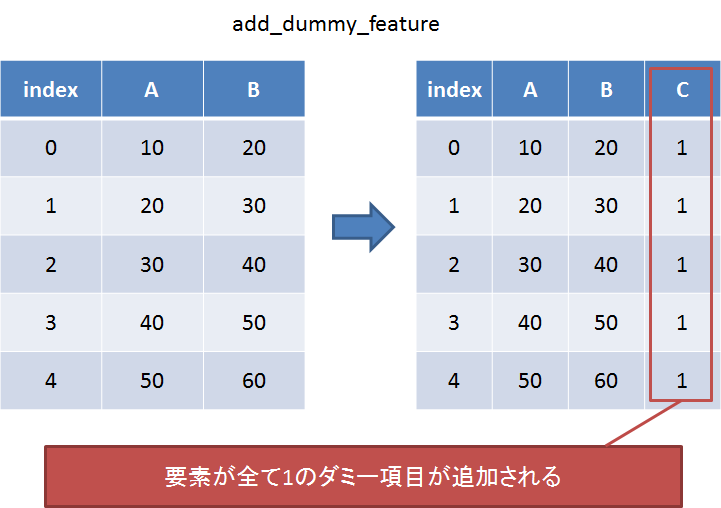
以下のように直接渡せば機能する。
sp.add_dummy_feature(df[df.columns[0:4]])
左の列にダミー配列が追加された。
Out[1]:
array([[ 1. , 5.1, 3.5, 1.4, 0.2],
[ 1. , 4.9, 3. , 1.4, 0.2],
[ 1. , 4.7, 3.2, 1.3, 0.2],
[ 1. , 4.6, 3.1, 1.5, 0.2],
[ 1. , 5. , 3.6, 1.4, 0.2],
[ 1. , 5.4, 3.9, 1.7, 0.4],
[ 1. , 4.6, 3.4, 1.4, 0.3],
...
binarize
閾値を設けてそれより大きい値を1に、小さい値を0にする(二値化)。
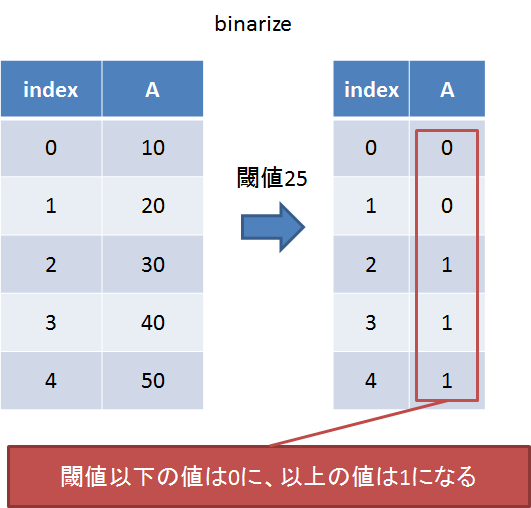
以下のように直接渡せば機能する。
sp.binarize(df[df.columns[0]], threshold=df[df.columns[0]].mean())
平均値以下の値は0に、それ以上の値は1に変換された。
Out[1]:
array([[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.,
0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.,
0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.,
0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 1., 1.,
1., 0., 1., 0., 1., 0., 1., 0., 0., 1., 1., 1., 0.,
1., 0., 0., 1., 0., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 0., 0., 0., 0., 1., 0., 1., 1., 1., 0., 0., 0.,
1., 0., 0., 0., 0., 0., 1., 0., 0., 1., 0., 1., 1.,
1., 1., 0., 1., 1., 1., 1., 1., 1., 0., 0., 1., 1.,
1., 1., 1., 1., 0., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 0.,
1., 1., 1., 1., 1., 1., 1.]])
Binarizer
基本的な機能はbinarizeと同様だが、パラメータ設定と適用を分けて実施することができるため、一度覚えさせたパラメータをほかのデータにも適用したいときに有効。
binarizer = sp.Binarizer(threshold=df[df.columns[0]].mean()) binarizer.transform(df[df.columns[0]])
結果はbinarizeと同様になる。
data
調査中。
FunctionTransformer
全要素にsin関数等の関数処理を一括適用したいときに使う。

①関数を渡す。②適用する。という2段階で使う。
transformer = sp.FunctionTransformer(np.sin) transformer.transform(df[df.columns[0]])
全ての値をsin関数に通した結果が得られた。
Out[1]:
array([[-0.92581468, -0.98245261, -0.99992326, -0.993691 , -0.95892427,
-0.77276449, -0.993691 , -0.95892427, -0.95160207, -0.98245261,
-0.77276449, -0.99616461, -0.99616461, -0.91616594, -0.46460218,
-0.55068554, -0.77276449, -0.92581468, -0.55068554, -0.92581468,
-0.77276449, -0.92581468, -0.993691 , -0.92581468, -0.99616461,
...
imputation
使わないメソッドと思われる。
Imputer
欠損値の穴埋め。平均か中央値か最頻値かで選ぶ。
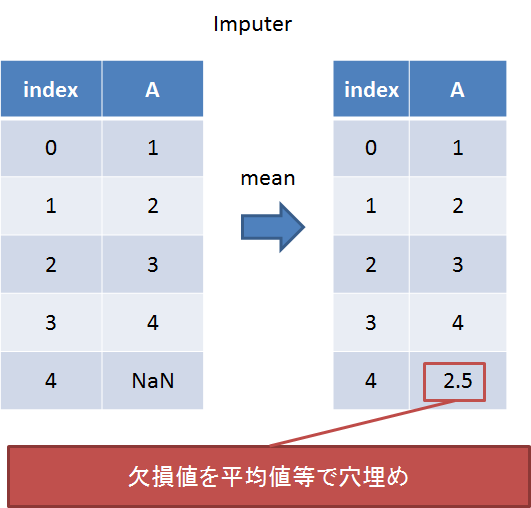
①欠損方法を指定する。②学習する。③適用する。という3段階で使う。
まずdfをtempにコピーし、1つ目のデータを欠損値にしておく。
temp = df.copy() temp.ix[0, 0] = np.NaN
確かに1つ目が欠損になっている。
temp[temp.columns[0]] Out[1]: 0 NaN 1 4.9 2 4.7 3 4.6 4 5.0 5 5.4 6 4.6 ...
次に欠損値にNaNを指定し、穴埋め方法として平均処理するように設定。
imp = sp.Imputer(missing_values='NaN', strategy='mean', axis=1) imp.fit(temp[temp.columns[0]]) imp.transform(temp[temp.columns[0]])
欠損値が平均値で埋められた。
temp[temp.columns[0]] Out[1]: 0 5.84832215 1 4.9 2 4.7 3 4.6 4 5.0 5 5.4 6 4.6 ...
KernelCenterer
写像関数。何のために使うものなのかは調査中。
①インスタンスを作る。②学習する。③適用する。という3段階で使う。
kc = sp.KernelCenterer() kc.fit(df[df.columns[0]]) kc.transform(df[df.columns[0]])
学習と適用を同じデータにすると全て同じ値になる模様。
Out[1]:
array([[ 870.65666667, 870.65666667, 870.65666667, 870.65666667,
870.65666667, 870.65666667, 870.65666667, 870.65666667,
870.65666667, 870.65666667, 870.65666667, 870.65666667,
870.65666667, 870.65666667, 870.65666667, 870.65666667,
...
fitするデータを変えてみた場合。
kc = sp.KernelCenterer() kc.fit(df[df.columns[1]]) kc.transform(df[df.columns[0]])
さっきとは少し違う結果になった。
Out[1]:
array([[ 453.85666667, 454.15666667, 453.75666667, 453.75666667,
453.65666667, 453.75666667, 453.45666667, 453.85666667,
453.75666667, 454.05666667, 453.95666667, 453.65666667,
454.05666667, 453.55666667, 454.05666667, 453.55666667,
...
グラフにしてみる。よく分からないが非線形に写像されているようだ。
Out[1]: plt.plot(df[df.columns[0]], kc.transform(df[df.columns[0]])[0], 'o')
label
使わないメソッドと思われる。
label_binarize
要素ごとに一致する値があるかどうかの列を作る。オブジェクト型の説明変数を機械学習にかけたいときによく使う前処理手法。
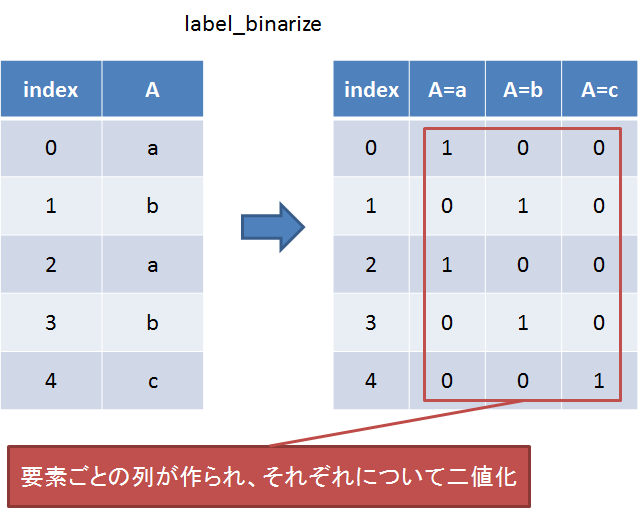
データと項目を渡すことで動作する。
sp.label_binarize(df[df.columns[4]], classes=df[df.columns[4]].unique())
[‘Iris-setosa’, ‘Iris-versicolor’, ‘Iris-virginica’]に一致するかどうかの結果が返される。
Out[1]:
array([[1, 0, 0],
[1, 0, 0],
[1, 0, 0],
[1, 0, 0],
[1, 0, 0],
...
LabelBinarizer
基本的な機能はlabel_binarizeと同様だが、パラメータ設定と適用を分けて実施することができるため、一度覚えさせたパラメータをほかのデータにも適用したいときに有効。
①インスタンスを作る。②classを指定する。③適用する。という3段階で使う。
lb = sp.LabelBinarizer() lb.fit(df[df.columns[4]].unique()) lb.transform(df[df.columns[4]])
結果はlabel_binarizeと同様になる。
LabelEncoder
文字列要素を数値コードに変換する。オブジェクト型の目的変数を機械学習にかけたいときによく使う前処理手法。
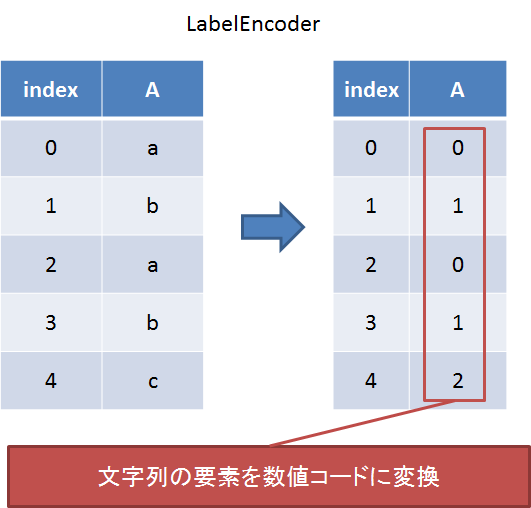
①インスタンスを作る。②classを指定する。③適用する。という3段階で使う。
le = sp.LabelEncoder() le.fit(df[df.columns[4]].unique()) le.transform(df[df.columns[4]])
[‘Iris-setosa’, ‘Iris-versicolor’, ‘Iris-virginica’]が[0, 1, 2]に変換された形で返される。
Out[1]:
array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2], dtype=int64)
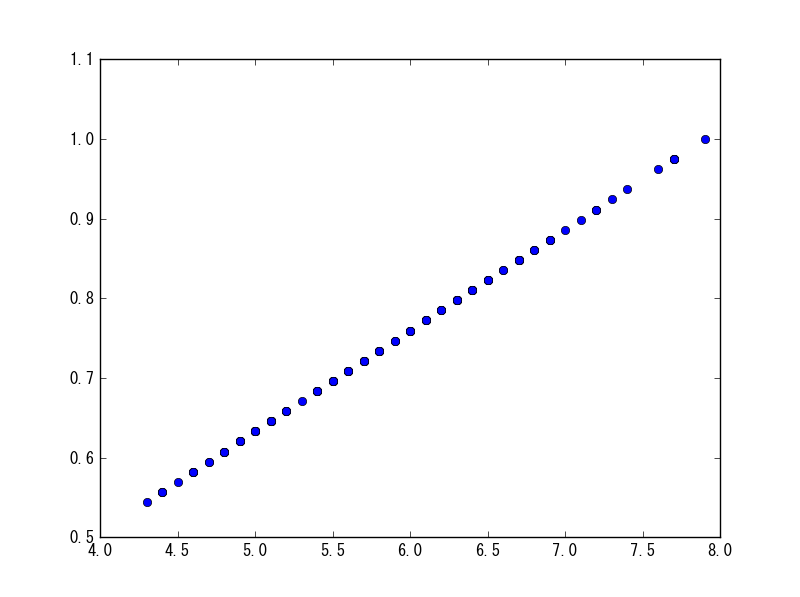
maxabs_scale
直接引数に与えれば、各列ごとにその列の最大値で割った値が返ってくる。
sp.maxabs_scale(df[df.columns[0]])
グラフにしてみると、確かに最大が1になっていることが分かる。
plt.plot(df[df.columns[0]], sp.maxabs_scale(df[df.columns[0]]), 'o')
MaxAbsScaler
基本的な機能はmaxabs_scaleと同様だが、パラメータ設定と適用を分けて実施することができるため、一度覚えさせたパラメータをほかのデータにも適用したいときに有効。
①インスタンスを作る。②学習する。③適用する。という3段階で使う。
mas = sp.MaxAbsScaler() mas.fit(df[df.columns[0:4]]) mas.transform(df[df.columns[0:4]])
結果はmaxabs_scaleと同様になる。
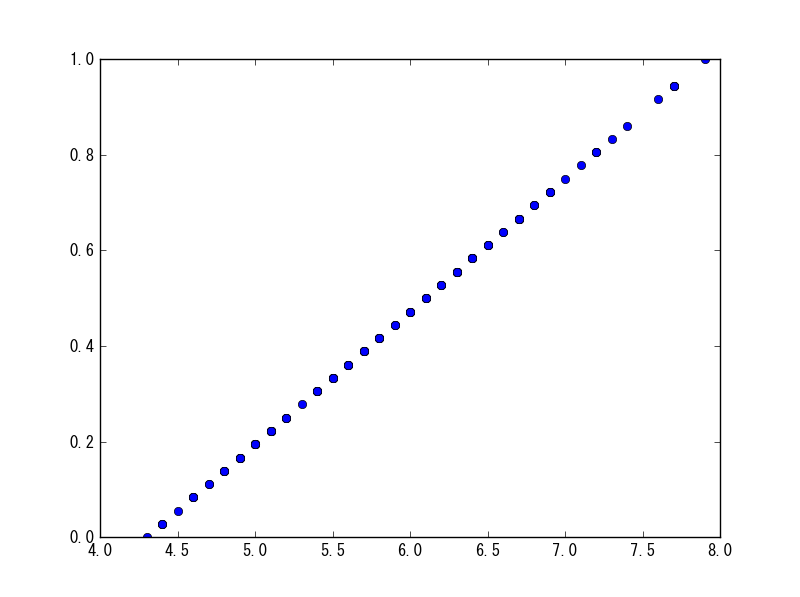
minmax_scale
直接引数に与えれば、各列ごとに最大値が1、最小値が0になるように線形写像された結果が返ってくる。
sp.minmax_scale(df[df.columns[0]])
グラフにしてみると、確かに最大が1、最小が0になっていることが分かる。
plt.plot(df[df.columns[0]], sp.minmax_scale(df[df.columns[0]]), 'o')
MaxAbsScaler
基本的な機能はminmax_scaleと同様。使い方はmaxabs_scaleに対するMaxAbsScalerと同様。
MultiLabelBinarizer
入れ子になっている要素列に対して使えるバージョンのLabelBinarizer。irisデータは該当しないため未検証。
normalize
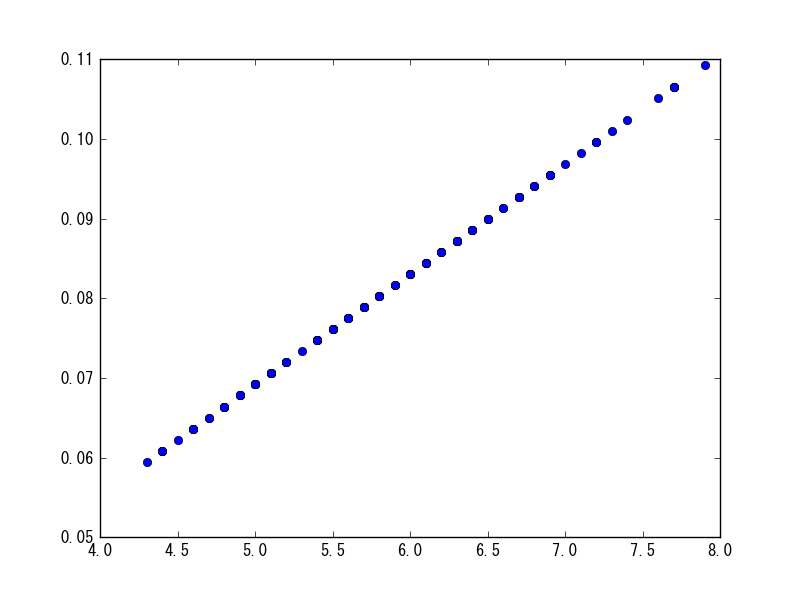
直接引数に与えれば、l1又はl2ノルムに基づき正規化された結果が返ってくる。
sp.normalize(df[df.columns[0]], norm="l1") plt.plot(df[df.columns[0]], sp.normalize(df[df.columns[0]])[0], 'o')
Normalizer
基本的な機能はnormalizeと同様。使い方はmaxabs_scaleに対するMaxAbsScalerと同様。
OneHotEncoder
調査中。
PolynomialFeatures
引数に対して、指定した次数までの多項値を作成する。例えば、引数にx,yを渡し、次数に2を指定した場合は以下のようになる。
[x, y] ⇒ [1, x, y, x*2, x*y, y*y]
使い方は①インスタンス作成、②適用の2段階が必要。
pf = sp.PolynomialFeatures(degree=2) pf.transform([2, 3])
すると以下のような結果が得られる。
Out[1]: array([[ 1., 2., 3., 4., 6., 9.]])
robust_scale
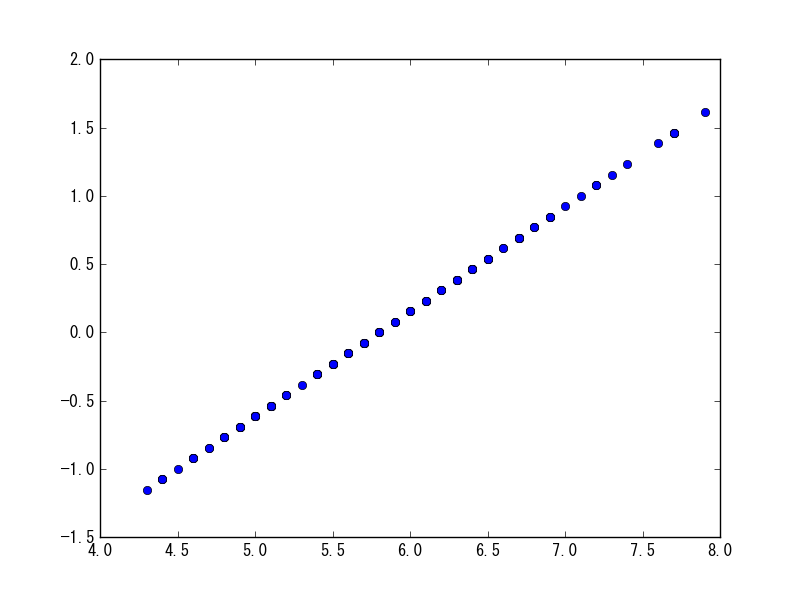
動作内容は調査中。何かしらの線形写像を実施している。
sp.robust_scale(df[df.columns[0]])
RobustScaler
基本的な機能はrobust_scaleと同様。使い方はmaxabs_scaleに対するMaxAbsScalerと同様。
scale
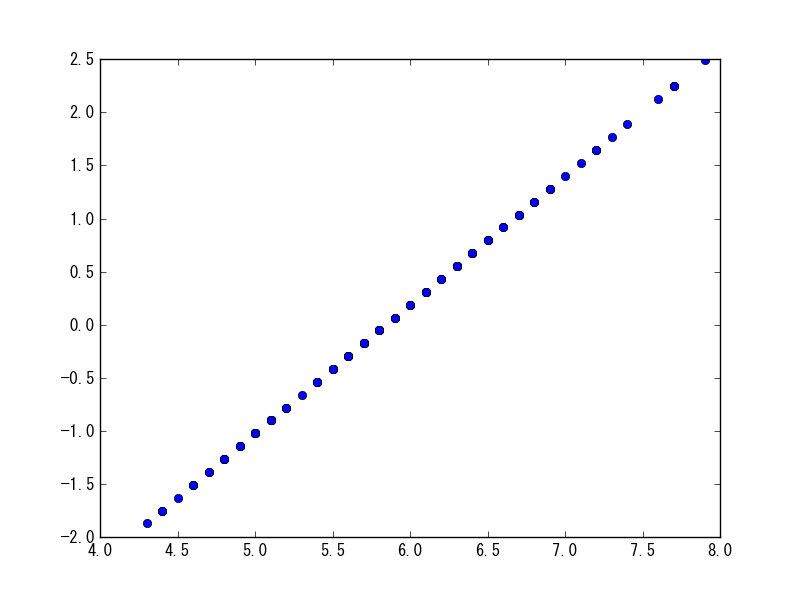
平均を0にし、標準偏差を1に設定する所謂正規化を実施したいときに使う。
sp.scale(df[df.columns[0]])
StandardScaler
基本的な機能はscaleと同様。使い方はmaxabs_scaleに対するMaxAbsScalerと同様。
まとめ
sklearnのpreprocessingは便利なものがあるんだろうな、と思いながら調べられてなかったので、この機会に(ほぼ)全メソッドについて紹介しました。いろいろなものがありましたが、とりあえずよく使うのは以下だと思います。
- 数値データの正規化:scale(又は、StandardScaler
- 数値データのフラグ化:binarize(又は、Binarizer
- 文字列(説明変数)のコード化:label_binarize(又は、LabelBinarizer
- 文字列(目的変数)のコード化:LabelEncoder